最近、ライフサイエンス分野のエピジェネティクスで目についた報告がありましたので紹介します。
■エピジェネティクスの話題:メッセンジャーRNAに付けられた新しいメチル化標識
Kietry & Kool、Nature、530,423-424,2016.
http://www.nature.com/nature/journal/v530/n7591/standfirst/530423a_ja.html
・メチル化はDNA並びにヒストンタンパク質で広く見つかっており、エピジェネティクス因子として、発生における細胞の分化決定段階等での作用が知られております。一方、メッセンジャーRNA(mRNA)では5メチルシトシン、6メチルアデノシンなどがあり、図1に示すRNAレベルでの動的で、可逆的な修飾によって転写後調節にかかわっており、N 6 -メチルアデノシン、即ちm6Aは、mRNAの局在、安定性、翻訳及びスプライシングに影響を与えているといわれております。
・N 1-メチルアデノシン、即ちm1Aは、mRNA損傷のマークとして、すでに知られており、酵素ALKBH3によって損傷mRNAを除いて、その毒性を減少させることが示されております。m1AはRNA分子をアルキル化剤に曝すことで形成され、ジムロート(Dimorth)転位によって容易にm6Aに変換されます(図1)。その上、m6AはmRNAに大量に存在しますが、一方でm1Aは少なく、これまで細胞性損傷のマーク以外での明瞭な役割は考えられていなかったようです。それには、m1Aが不安定で、容易にm6Aへ変換されてしまい、定量的な測定ができていなかったことにも一因があったようです。
・最近、シカゴ大のDominissini*1らはmRNAのメチル化修飾として、N 1 -メチルアデノシン(m1A)が、生物学的プロセスで新しい機能として、細胞性ストレス応答やシグナリングプロセスでの役割を担って1る細胞性のスイッチとしての可能性が示唆されております。
*1;Dominissini et.al., Department of Chemistry and Institute for Biophysical Dynamics, The University of Chicago, USA
Nature 530,441-446,2016.
http://www.nature.com/nature/journal/v530/n7591/full/nature16998.html
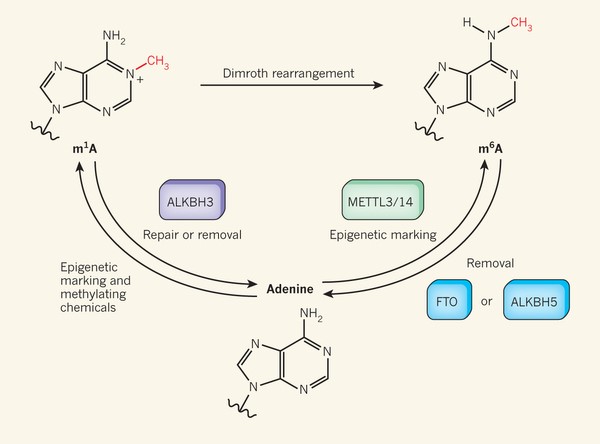
図1、アデニンの動的メチル化;
・RNA分子中のアデニン塩基のN1位のメチル化は、N1メチルアデノシン(m1A)修飾を産生する。これは化学的メチル化によって生じる損傷の結果として起こると考えられている。
・Dominissini et al.らの報告;損傷されていないメッセンジャーRNA分子に(m1A)の存在を報告し、「細胞性エピジェネティクなマーク」を表現していると提唱している。RNA損傷に応答して、修復酵素ALKBH3は塩基を修復するために、このメチル基を除去することができる。しかし、この酵素、或は別酵素がエピジェネティクなマークを除去するために機能だけでなく、どのようにこのマークが付け加えられるのかは、はっきりしていない。別型のメチル化アデニンm6AはMETTL3/14 によって付け加えられ、FTO or ALKBH5によって除かれるmRNAのエピジェネティクなマークであることは既に認識されている。
・アルカリ性の条件下で、m1AはDimroth 転移によってm6Aに変換される。
◆細胞内代謝の調節に関する新しいスイッチとしてのメチル化マーク(m1A)?;
―mRNAのアデニンN1位のメチル化(m1A)が細胞内タンパク質レベルを調節?―
・今回、Dominissini*1らは、m1Aからm6Aへの変化を10%以内に抑えるm1Aの化学的不安定性を抑制する方法をみつけ、質量分析(Mass Spectrometry)によってm1Aを正確に定量化することを可能にした結果、m1Aの構造的な特徴がいくつか見いだされました。m1AはmRNAの第1スプライス部位上流の開始コドン周辺に豊富にあり、その分布はGCリッチ領域の翻訳開始部位周辺など、より構造性の高い領域にあります。m1AをもつmRNAには、m1Aを持たないものに比べて、タンパク質レベルが1.7倍と高いものがあり、m1Aを含むmRNAの割合が遺伝子発現レベルと正の相関することから、スプライシングバリアントなどを経て成熟mRNAになって、最終的に細胞内タンパク質レベルとなる転写・翻訳プロセスでの調節に関与しているかもしれないと説明しています。構造的には、m1Aは中性pHで正電荷をもち、二重らせん対合をブロックするので、タンパク翻訳開始位置での塩基対形成、RNAフォールディングや静電気的相互作用に影響を与えているかもしれません。
m1Aは酵母から哺乳類の真核細胞までの遺伝子の転写産物に見られ、ヒト平均では、転写物の20%と推定されています。この特徴はマウスとヒト細胞で高度に保存され、メチル化mRNAの翻訳促進においてm1Aが機能的役割を担うことが強く示唆されております。
以下、Dominissini*1らの報告記載の図の主な説明;
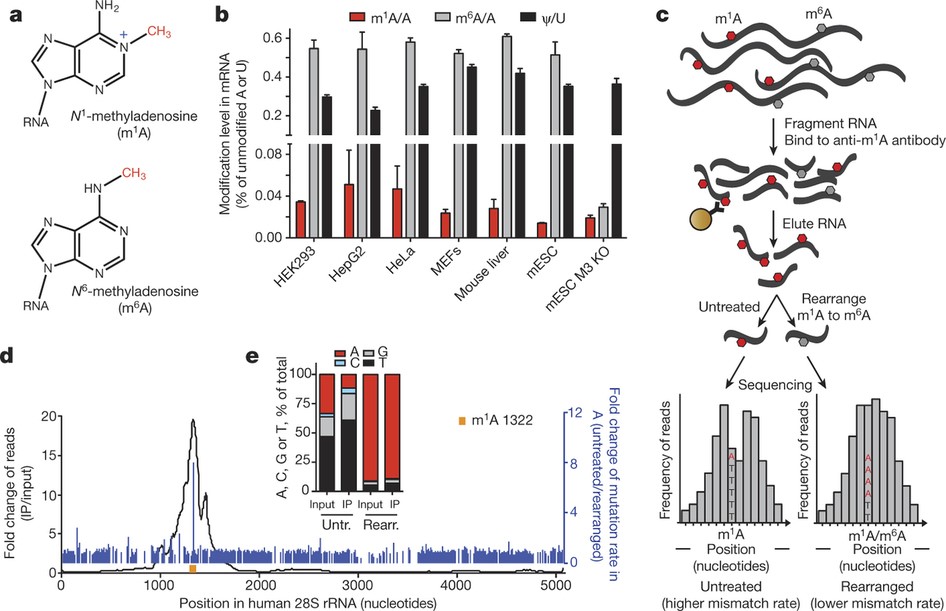
図2a;m1A 及びm6Aの化学構造を示す。
図2b;ヒトとマウス分離細胞から得られたm1A、m6AのLC-MS/MS定量;各修飾ヌクレオシドは未修飾に対する%で表示。
図2c;m1A配列の模式的アウトライン;RNAをフラグメント化し、抗m1A抗体で免疫沈降させ、回収したRNAフラグメントをcDNAに変換して配列決定、或はcDNA合成前に部分的にm1A-to-m6A変換をするよう処理。
図2d;既知のヒト28S、rRNAのm1A配列(黒曲線)。rRNAのm1A 1322部位の検出。
図3a;メチル化転写物の代表的なプロット;免疫沈降物(m1A IP;赤)。青の標準化配列補てんはUCSCフォーマットの遺伝子構築上に示してある。
図3c;Hela細胞;発現レベルの上昇とともにメチル化遺伝子が増加。
図2、哺乳類のmRNAで新たに同定したm1A 配列の構成マップ;Development of m1A-seq to map a newly identified constituent of mammalian mRNA.
a, Chemical structures of m1A and m6A. Methyl groups (-CH3) are in red and the positive charge (+) on m1A is in blue. b, LC-MS/MS quantitation of m1A, m6A and Ψ in human and mouse mRNA isolated from the indicated cell types. The level of each modified nucleoside is indicated as a percentage of the unmodified one. Mean values ± s.e.m. are shown, n = 3. MEFs, mouse embryonic fibroblasts; mESC, mouse embryonic stem cells; M3 KO, Mettl3 knockout. c, Schematic outline of m1A-seq. RNA is fragmented and subjected to immunoprecipitation using anti-m1A antibody. Eluted RNA fragments are converted to cDNA and sequenced, or treated to induce partial m1A-to-m6A rearrangement before cDNA synthesis. m1A causes both reverse transcription stops and read-throughs accompanied by mismatches, to produce typical peaks with a central trough and an adenosine with increased mismatch rate (left). Partial rearrangement of m1A to m6A attenuates the effect (right). m1A is in red, m6A is in grey; mismatch rate is illustrated as the ratio between A (red) and T (black). d, Detection of the known m1A 1322 site (yellow) in human 28S rRNA validates m1A-seq (black curve). High fold-change in mismatch rate (before and after m1A-to-m6A rearrangement in immunoprecipitation, blue lines) independently identifies m1A 1322 with single-nucleotide resolution. e, Mismatch rates in m1A 1322 increase after immunoprecipitation and decrease upon rearrangement.
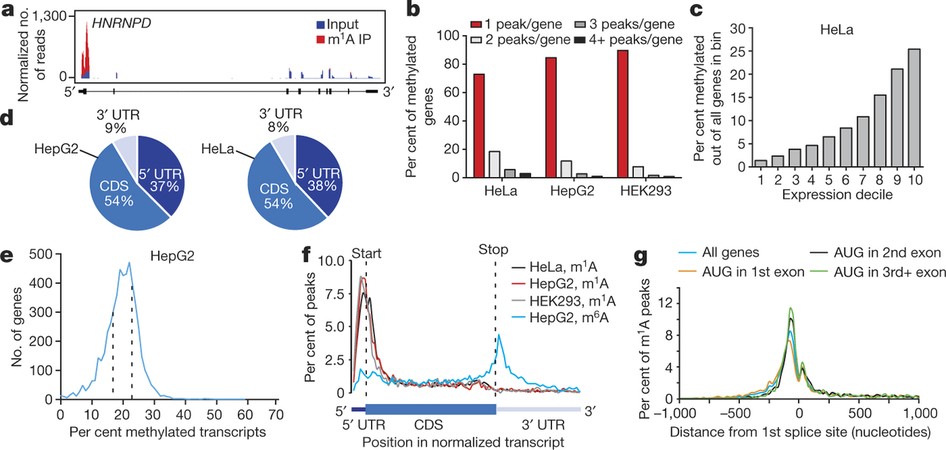
図3、m1A is associated with translation initiation sites (TISs) in the human transcriptome.
a, The percentage of methylated genes that carry 1, 2, 3 or 4+ peaks per gene in the indicated mouse cell types. Most genes carry only one m1A peak. b, The percentage of methylated genes in mouse liver cells increases with expression level. c, Pie chart presenting the fraction of m1A peaks in each of three non-overlapping transcript segments (5′ UTR, CDS and 3′ UTR) in mouse liver. d, Metagene profiles of m1A peak distribution along a normalized transcript composed of three rescaled non-overlapping segments illustrated below, in the indicated mouse cell types. m1A peaks cluster around the AUG start codon. e, Metagene profiles of mouse m1A peak distribution in a non-normalized window centred on the nearest splice site. Peaks are sorted by the exon containing the AUG codon in that gene. f, Length-adjusted minimum free energy (aMFE) for 5′ UTRs of methylated, unmethylated and all genes in mouse liver. Box limits and whiskers are as indicated in Fig. 3b. P values are indicated, t-test. g, Human–mouse m1A conservation expressed as per cent of orthologous positions with shared m1A peaks according to their location in the transcript.
以上