今回はDNAの配列決定法であって、第3世代技術の1つであるDNA合成による配列決定法(SBS法;DNA sequencing by synthesis)についてのPNSA論文1)を紹介します。
■DNA合成に基づく、ナノポアアレイによる塩基配列の決定法
Fuller CW, et.al.,;Real-time single-molecule electronic DNA sequencing by synthesis using polymer-tagged nucleotides on a nanopore array.
PNAS 113 (19) 5233-5238,2016.http://www.pnas.org/content/113/19/5233.full
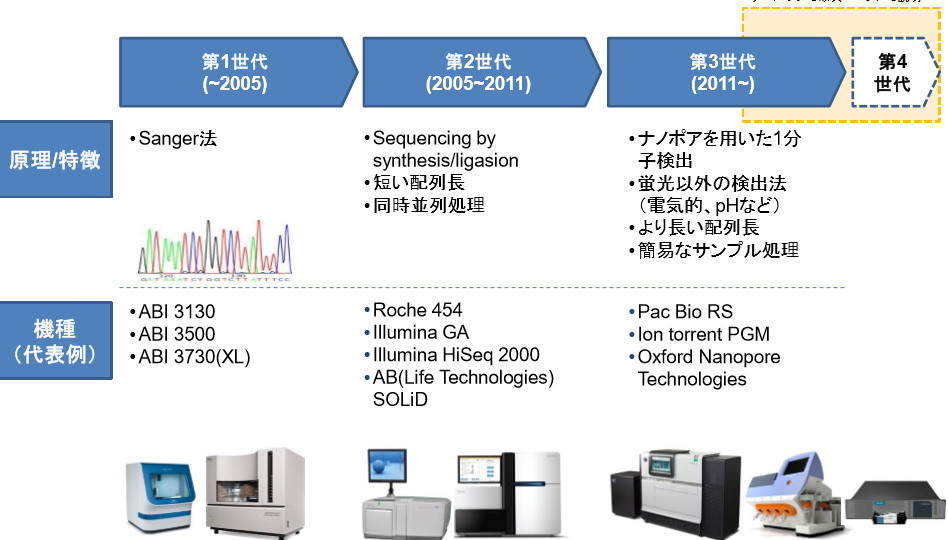
・DNAの塩基配列の決定に関する技術は、この40年間で著しく進展し、生物学は言うに及ばず、個別化医療「Precision medicine(Personalized medicine)」の領域でも、極めて重要な技術と認識されています。第1世代からすでに第3世代へと技術が進展してきており(下図)、今回紹介する、このナノポアを利用したSBS技術は第3世代に分類されています。
*SCIENTIFIC REPORTS,2 : 501, 2012.(July)。http://www.ikegin-c.jp/pdf/frm1304254.pdf(Quantum Biosystems Inc)
<前置き>
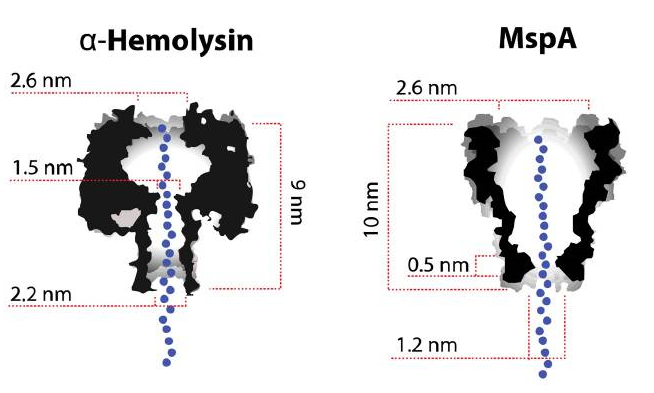
・ナノポアは生体膜あるいは人工的脂質二重膜に在るナノメートル(10-9)レベルの穴(ポア)のイオンチャネルであり、このナノポアを通るイオン電流の測定は神経生物学や生物物理学では一般的です。黄色ブドウ球菌のたんぱく質αへモリシンや、マイコ科のスメグマ菌のMspAなどの比較的大きなイオンチャネル(図1)は長時間、開口したままに出来るので、膜チャネルを通過する連続的なイオンの流れを可能にしています。
・αへモリシンは7量体でナノポアを形成しており、このナノポアを含む膜間に電極対を置くと、長く伸びた1本鎖DNAを通すことができ、イオン電流が測定できます。しかもナノポアを通過するDNAや各核酸塩基の種類によってイオン電流量が変化する為、その違いを測ることによって、塩基配列を決定することが可能となります。
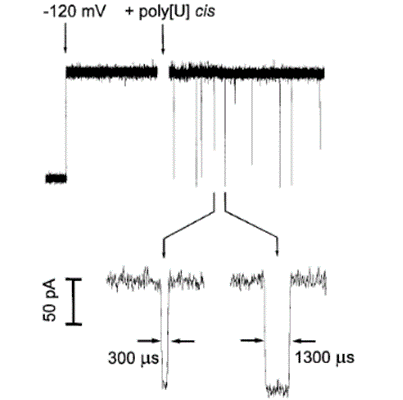
・ナノポアを配列決定に利用する研究の実用化には、これまで20年間かかっており、1996年にKasianowicz1)やGundlach2)達はナノポアを通るイオン電流を観測(図2)1)しております。図2、矢印でナノポアを埋め込んだ膜に電圧120mVかけると、120pAの連続的な電流が生じます。単一分子のpoly[U](平均210塩基)のオリゴマーは、αへモリシンチャネルのイオン電流を一時的に封鎖(ブロック)します。このオリゴマーによるイオン電流の短時間ブロックは、スパイク様の単線で観測され、拡大図にその典型的な300μsあるいは1300μsのイオン電流ブロックがみられます。
図1;天然のナノポア、αへモリシンとMsp
 http://www.ncbi.nlm.nih.gov/pmc/articles/PMC3804109/figure/F2/
http://www.ncbi.nlm.nih.gov/pmc/articles/PMC3804109/figure/F2/
1、Kasianowicz et al.;This process can be observed at the single-molecule level by monitoring the ionic current flowing through the pore. PNAS 93:13770-3,1996
図2;単一分子のナノポアイオン電流測定
2、Butler TZ, Gundlach JH, MABiophys J. Determination of RNA orientation during translocation through a biological nanopore. Biophys J. 90:190-9. 2006.
◆これまでに実用化に向けたイオンチャネリング技術には主に次の3つの方法が報告されています。
① Strand sequencing(SS)法1,3);ナノポアを連続して通過する核酸の配列を同定する。
② Exonuclease-based nanopore sequencings(EBS)法4);エキソヌクレアーゼによるナノポアシークエンシング法で、DNA分子から1個づつ核酸が酵素的に切断され、ナノポアを通って捕獲・パスされてモニターされる。
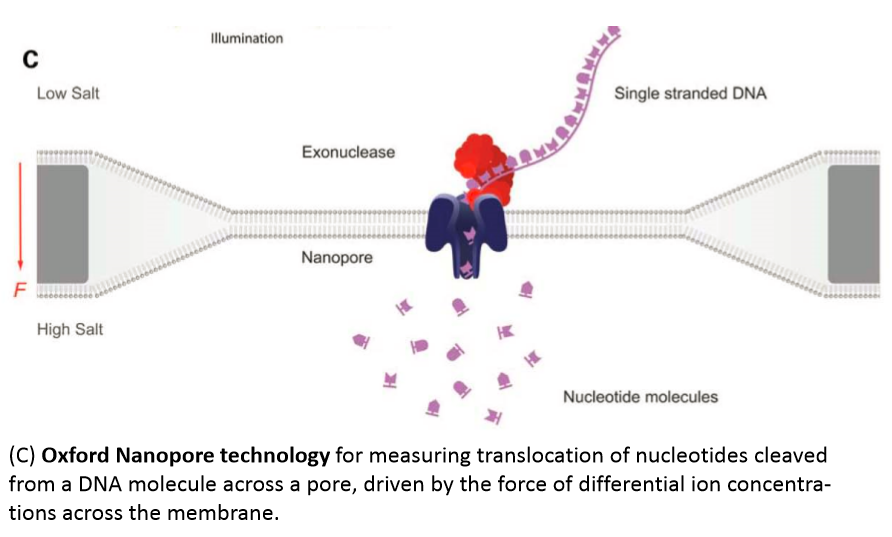 *Oxford NanoporeのStrand Sequencing原理;脂質2重膜にタンパク質ナノポアを埋め込んだチップから、市販版はポリマー膜にタンパク質ナノポアを埋め込んだチップ“MinIONデバイス”でシーケンシングする。http://bioinfo.ut.ee/JC/2010-2011/ulvi_jc_2010_1.pdf
*Oxford NanoporeのStrand Sequencing原理;脂質2重膜にタンパク質ナノポアを埋め込んだチップから、市販版はポリマー膜にタンパク質ナノポアを埋め込んだチップ“MinIONデバイス”でシーケンシングする。http://bioinfo.ut.ee/JC/2010-2011/ulvi_jc_2010_1.pdf
③ ナノポアSBS(nanopore SBS;DNA sequencing by synthesis)法5、6):ポリマータグを核酸に結合させた化合物を基質として用い、ポリメラーゼ酵素と融合させて一体化したナノポアをイオン電流の測定に用いた方法で、今回、紹介する技術。
・これらの方法すべてにおける重要なポイントは、解読する各塩基配列の順序に誤りがなく、かつ正しく決定することです。核酸などの化合物が比較的速くナノポアを通過するので、通過速度をゆっくりとさせる工夫が要り、ポリメラーゼの反応速度を正確にコントロールことが要求されます。このため、①SS法ではチャネル内の核酸の複数性を解読する必要があり、通過速度をスローダウンする必要があります。②EBS法ではイオン電流シグナルが有効に得られるような酵素によって、放出された核酸がナノポアに密接で、かつゆっくりした速度を保証する必要があります。エキソヌクレアーゼはDNAの二次構造とその配列に敏感で速度が変わりますが、③で使用するDNAポリメラーゼは、そうした影響を受けにくい酵素です。
・今回紹介する本論文の③SBS法は、コロンビア大学のFullerらによる報告です。核酸に結合したポリマータグがナノポア通過時間をスローダウンさせることができ、しかも似たような2個のピリミジン核と2個のプリン核の核酸を、そのポリマータグの分子構造に変化を付けることで、4種の核酸が明確に識別することができる、シークエンシング技術です。
===================================
3)Maitra RD, et.al.,Recent advances in nanopore sequencing. Electrophoresis 33(23):3418–3428, 2012.
4)Clarke J, et al.;Continuous base identification for single-molecule nanopore DNA sequencing. Nat Nanotechnol 4(4):265–270, 2009.
5)Kumar S, et al.;PEG-labeled nucleotides and nanopore detection for single molecule DNA sequencing by synthesis. Sci Rep 2:684, 2012.
6)Fuller CW, et.al., Real-time single-molecule electronic DNA sequencing by synthesis using polymer-tagged nucleotides on a nanopore array.PNAS 113 (19) 5233-5238,2016.
*参考;
・Andrew H Laszlo (Jens H Gundlach)Decoding long nanopore sequencing reads of natural DNA;Nature BiotechnologyVolume:、32,Pages:829–833,Year published:2014.
・ゲーティングナノポア技術を使ったDNA解析;2009年にナノポア技術を用いたDNA解読で世界初。OhshiroT(Kawai);Single-Molecule Electrical Random Resequencing of DNA and RNA;Scientific Reports 2, 501 (2012)
■DNA合成に基づく、ナノポアアレイによる塩基配列の決定法
著者;Fuller CW, et.al.,
所属;Center for Genome Technology and Biomolecular Engineering, Columbia University, New York, NY 10027
タイトル;Real-time single-molecule electronic DNA sequencing by synthesis using polymer-tagged nucleotides on a nanopore array.
論文;PNAS 113 (19) 5233-5238,2016.
・DNAシークエンシングは新しい個別化医療(precision medicine)において、重要な役割を担う技術となってきている。ゲノム、エピゲノム情報は、疾患の性質、臨床的予後、治療法などにおいて、日常的ルーチン化するためにシークエンス精度を上げて、解読する長さを増加させ、スピードアップし、コストをさげるなど、もう1オーダー程度下げる必要があると著者らは言及しています。
◆合成ポリマータグを結合しているDNAポリメラーゼの核酸基質;
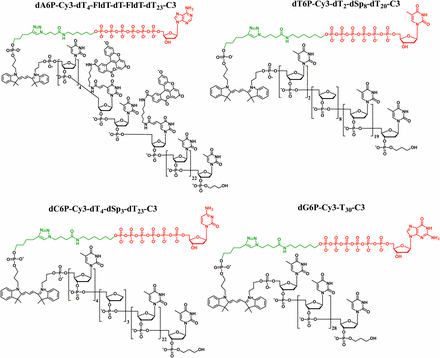
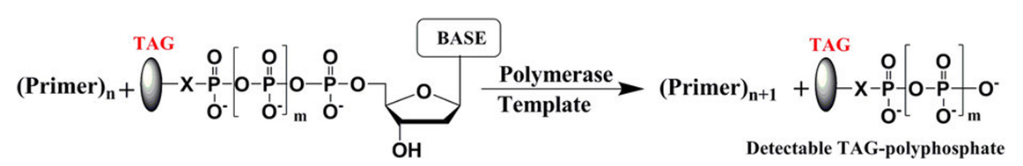
本論文でコロンビア大の研究者たちは、ナノポアに流れる電流をブロックできるポリマータグをデザインし化学合成した。図3に示すように、4種類のDNAポリメラーゼの基質となる核酸ポリマーは、すべて末端に基質となる核酸(赤)部位を配しており、リボース核の5‘位に6個のリン酸基(赤色)を繋げ、さらに共通リンカー(緑色)を介してデオキシヌクレオチド核酸(dT基)30個を配置し結合させている。その特徴は、ポリマータグであるdT基を化学修飾してポリマー構造に変化を持たせ、4種類の核酸基質が異なるようにしている点にあります。
図3;ポリマー・タグ化した核酸の構造;
図3;ポリマー・タグ化した核酸の構造;各核酸(赤)の5‘側に6個のリン酸基(赤色)を繋ぎ、さらに共通リンカー(緑色)を介して、dTを主に4種類の核酸;A;dA6p、T;dT6p、C;dC6p、G;dG6pを合成。構成;A;dT27+FL/dT/FLdT=dT30、T;dT22+dsp8=dT30、C;dT27+dsp3=dT30、G;dT30、ポリマー長は30個の主にデオキシ核酸からなる。
図4;ナノポアSBSの原理;
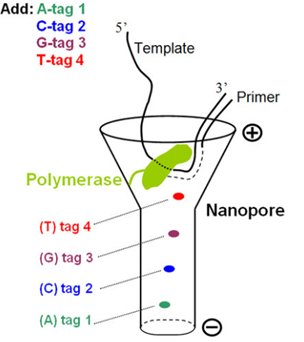
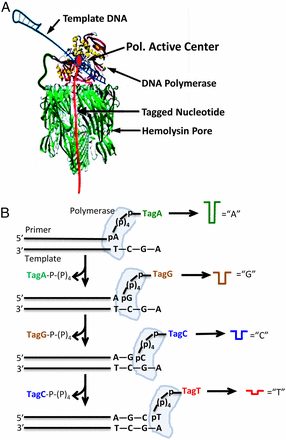 図4;ナノポアSBSの原理;(A)ナノポア・ポリメラーゼの配列決定エンジン;DNAポリメラーゼ分子1個が共有結合でαHLヘプタマー(7量体)で構成されているナノポアに付いている。プライマーとテンプレートDNA(二重ヘアピン配座)は、タグ化された核酸とともにポリメラーゼと複合体を作っている。(B)SBS模式図;ナノポアによる配列捕獲とタグ化ヌクレオチド核酸の検出、ポリメラーゼ反応によるDNA鎖の伸長とともに、ヌクレオチド核酸に固有の電流ブロックが生じる。α-HL;α-Hemolysin
図4;ナノポアSBSの原理;(A)ナノポア・ポリメラーゼの配列決定エンジン;DNAポリメラーゼ分子1個が共有結合でαHLヘプタマー(7量体)で構成されているナノポアに付いている。プライマーとテンプレートDNA(二重ヘアピン配座)は、タグ化された核酸とともにポリメラーゼと複合体を作っている。(B)SBS模式図;ナノポアによる配列捕獲とタグ化ヌクレオチド核酸の検出、ポリメラーゼ反応によるDNA鎖の伸長とともに、ヌクレオチド核酸に固有の電流ブロックが生じる。α-HL;α-Hemolysin
◆ナノポアSBSの原理;
・DNAポリメラーゼ(Phi29由来のDNApol)はαーヘモリシン(αHL)のナノポアに結合しており、リコンビナント融合タンパク質として一体化しています。このDNApolはポリマータグを含む核酸基質および標的テンプレートとタイトな複合体(ternary complex)を形成します(図4)。DNA合成によってDNA鎖が伸長するたびに、核酸が加水分解して遊離するポリマータグはナノポアに入って、イオン電流をブロックする(図4)。このブロックによってイオン電流が変化するので、変化量を測定し、モニターすることで、配列が決められます。
・この反応ステップを繰り返すことによって、テンプレートに沿ったDNA合成による伸長とともに、対応して連続して変化するイオン電流のブロック量が各核酸のポリマータグに応じて、識別できるので、各核酸による配列が決定できることになる。この方法は、タグ検出、すなわちヌクレオチドのDNAへの取り込み速度がナノポアによって捕獲されるポリマータグの結合速度より遅いので、電流ブロックとして測定され、達成できている。本研究での基質の修飾は、αHLチャネルの狭い構造近くに位置づけるようにし、ナノポアを通るイオン電流に直接影響することができるのです。
◆ナノポアを有するアレイチップ;
脂質二重膜にナノポア(αHL)をもつ264個の銀電極アレイチップ(図5)でシークエンシング反応をおこなう。そのチップ上に簡単なフローセルを組み立て、コンピュータ制御のシリンジポンプによって試薬は供給される。
図5; ナノポアSBSのCMOSチップ;
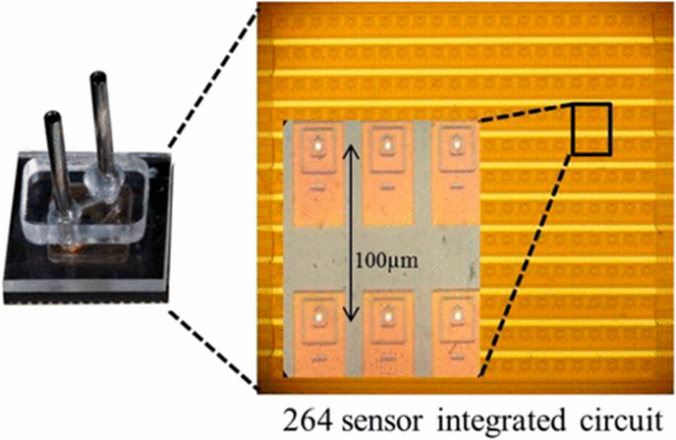 ・CMOSチップ*(~1x1㎜)は264個の銀電極アレイ(直径5μm)をもつ積層サーキットからなる。
・CMOSチップ*(~1x1㎜)は264個の銀電極アレイ(直径5μm)をもつ積層サーキットからなる。
・チップはアナログ・デジタル変換によって、全電極から独立に、1秒間に1000以上の速度で電気的測定を報告する。
・チップ(右図)を取り囲んでいるフローセル(左図)と試薬のインプットとアウトプットを含む。
・挿入図は電極アレイと個々のセンシングセルの構造詳細の拡大を示す。
*COSM;complementary metal-oxide semiconductor chip(金属半導体チップ)
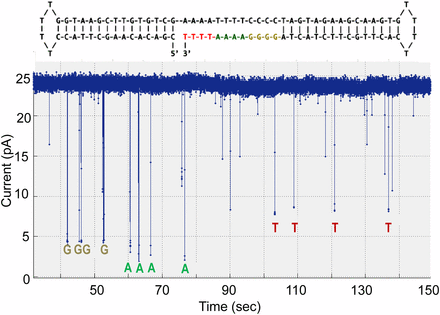
・図6、図7に実験結果を示す。ブロックされたイオン電流の割合は、各ポリマータグの立体構造的「バルキー」によって異なり、最もバルキーなタグである核酸分子;dA6p-Cy3‐dT4-FIdT-dT-FIdT-dT23‐C3は最も低い電流値~2pAを示し、未修飾のデオキシ核酸dTを30個もつタグの核酸dG6p-Cy3‐dT30‐C3は、~4pAを示した。 一方、低いバルキータグを有する核酸dT6p- Cy3‐dT2-dSp8-dT20‐C3と核酸dC6p- Cy3‐dT4-dSp3-dT23‐C3では、それぞれ~7PA及び~8PAの高い電流値が観察された。
図7にみられるように、連続して何回も捕獲された現象(スパイク様のシグナナルピーク)は、テンプレート配列の次の核酸が来るまで、続いてみられる。このイオン電流をブロックしたレベルでみられるシャッター現象は、ナノポアにポリマータグが取り込まれる前、ポリメラーゼへの結合とリリースを繰り返していることを反映していると考えられる。事実、図7に示すように実験条件を変えることで、このシャッター効果は低下できている。
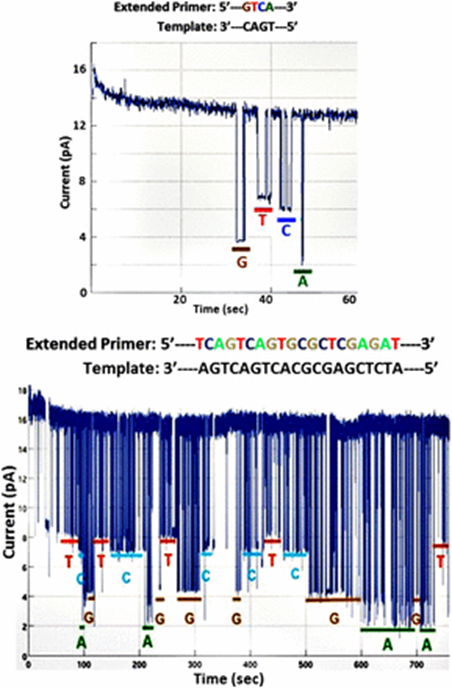
図6;ナノポアアレイチップによるSBSのシークエンシング
図6;αHLのナノポアに結合しているPhi29DNAポリメラーゼ分子、テンプレート、4種のタグ化ヌクレオチドの組み合わせによるシークエンシング。上図;テンプレートの長さ/4ヌクレオチド;4種類の塩基が明瞭に識別されている。trans膜側;3mMSrCl2、cis 膜側;0.1mMMnCl2。下図;テンプレートの長さ/20ヌクレオチド・長い配列を解読。trans膜側;3 mM SrCl2, cis 膜側;3 mM MgCl2, 0.7 mM SrCl2,
図7;ナノポアアレイチップによるSBSのシークエンシングSBSのシークエンシング結果;
図7;12塩基のDNAホモポリマー領域(色付き)におけるナノポアSBSの結果;図中の電流トレースはノイズレダクションをしていない生データのアウトプットを示している。
・図7の実験では、テンプレート3’末端がナノポアへ進入するのを防ぐために、二重のヘアピンプライマーを使用し、3‘末端から下流へ直ちにエンコードされて5′-GGGGAAAATTTT-3′配列をシークエンシングしている。ポリメラーゼ反応の間、わずかな電流低下はナノポア内でのタグ捕獲の指標であって、4つのポリマータグの構造差異の特徴的偏移の深さを伴っている。図中の他の偏移は、中心からわずか(<10ms)でノイズとして無視できるレベルにある。
(本論文に記載ない推測)ドライビングフォースはナノポアを含む膜間に電圧を印加することで、電位勾配(シス側‐、トランス側+)ができ、トランス側のSr2+カチオンがナノポアを通ってシス側に移動する際にイオン電流が発生する。一方、シス側にあるポリマータグはDNAポリメラーゼによってDNA合成の進行によって、放出され、その32個のリン酸基のO-アニオンの負電荷がトランス側に引き付けられて移動する際にイオン電流をブロックすることになる。
・今回のナノポアによるSBSシークエンシング法は明瞭な配列依存性の電気シグナルを分子レベルで連続的に検知できるシステムが、試薬の添加や除去をすることなく、短時間に達成できているとしている。
・図6に比べて図7の結果が示すように、イオン電流ブロックの帯状のスパイク様ピークがシヤープな単一バンドに変化している。しかしながら、各ピークの出現時間がまだ不均一で、かつ同定されていない予測以外の低いピークもみられており、改善の余地があるものと思われる。
・著者らも述べているように、今後の課題として、シークエンシングの速度、およびオーバーオール配列の正確さの確立が、さらに必要だとしており、他のポリメラーゼの検討や、識別のよいタグ化ポリマー核酸を開発するなど、システムにおけるパラメーターのさらなる改良が必要だと考えている。
◆参考;
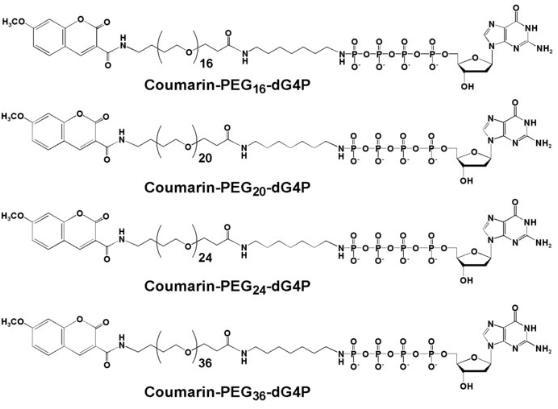
・最初は、ポリマータグとして、PEGの長さを変化させて核酸の基質を合成して検討、今回は、デオキシリボ核酸ポリマー型の核酸基質を開発。
・PEGタグの長さに応じて、ナノポアのイオン電流は低下するが、膜移動(Translocation)時間が異なること、およびPEGタグの合成過程に由来する高塩濃度がポリメラーゼ反応に良くなく、PEGに代わるデオキシリボ核酸のポリマーを開発した。この基質はヌクレオチド3リン酸(NTP)よりも優れた基質活性を示した。
*タグとしてPEGを使用したナノポア(nanopore)SBS法のDNAの塩基配列の決定;Kumar S, et al.;PEG-labeled nucleotides and nanopore detection for single molecule DNA sequencing by synthesis. Sci Rep 2:684, 2012.
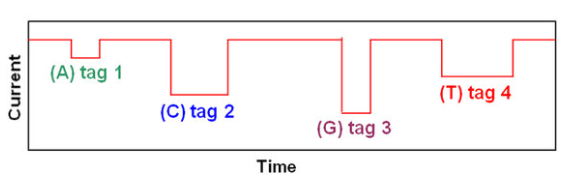
図;模式図;
SBSの間、5‘末端に結合していたポリマータグが放出されて、ナノポアに一度に1回入るたびにイオン電流がブロックされて変化している様子。
・各PEGポリマータグ結合した核酸塩基;
以上